Monitoraggio SARS-CoV-2 in acque reflue urbane

Il virus SARS-CoV-2 è emesso prevalentemente con le feci dal 70% delle persone infette a,b, sia che esse presentino sintomi evidenti da COVID-19, sia che siano parzialmente o completamente asintomatiche. Analizzare le acque in ingresso ai depuratori significa pertanto poter monitorare la presenza del virus di un determinato bacino, servito dal depuratore. Il vantaggio della metodologia consiste nel fatto che un semplice campione di acqua reflua rende possibile monitorare la diffusione del virus tra la popolazione in una determinata zona in maniera anonima, non invasiva e in modo efficiente e con costi ridotti.
Il monitoraggio
A partire da settembre 2021, il Laboratorio biologico partecipa al progetto di sorveglianza epidemiologica di SARS-COV-2 attraverso le acque reflue urbane (SARI, Sorveglianza Ambientale Reflue in Italia) e segue un protocollo nazionale per estrarre dalle acque reflue in ingresso ai depuratori il materiale genetico del Coronavirus SARS-CoV-2 e ricercarne frammenti genici. Il tutto è finalizzato al monitoraggio preventivo sulla presenza del virus e la sua possibile propagazione. I campioni vengono prelevati 2 volte a settimana presso 9 depuratori della provincia. L'estratto di ciascun depuratore viene inviato una volta al mese all'Istituto Superiore di Sanità di Roma per la ricerca delle varianti del SARS-CoV-2. I risultati delle analisi vengono pubblicati ogni mese sul sito dell'Istituto Superiore di Sanità. Il monitoraggio viene svolto in collaborazione con i gestori degli impianti di depurazione, l’Azienda Sanitaria dell'Alto Adige e gli uffici dell'Agenzia Provinciale per l'Ambiente e la Protezione del Clima.
Leggi anche l'articolo "Il monitoraggio del Sars-Cov-2 nelle acque reflue urbane" nella rivista "Ecoscienza" (n. 2, anno 2022).
Tra le persone infettate dal virus, solo quelle sintomatiche si rivolgono di norma al sistema sanitario per sottoporsi a test e vengono pertanto notificate, mentre molte persone con sintomatologia lieve o assente sfuggono al sistema di tracciamento. Tutti però fanno uso dei servizi igienici, e quindi qualsiasi emissione di virus finisce nelle acque reflue e attraverso il sistema fognario viene conferito ai depuratori. Analizzare le acque in ingresso ai depuratori significa poter monitorare la circolazione del virus.
Il prelievo dei campioni
Il prelievo dei campioni avviene 2 volte a settimana: lunedì (raccolta delle 24 ore da domenica mattina a lunedì mattina) e giovedì (raccolta delle 24 ore da mercoledì mattina a giovedì mattina). Il campione viene formato raccogliendo quantitativi di refluo nel corso delle 24 ore in maniera proporzionale all’afflusso orario; in questa maniera il campione è rappresentativo di tutto il bacino d’utenza dei singoli depuratori.
L’Ufficio Tutela acque ha individuato 9 depuratori rappresentativi del territorio, che tenessero conto anche delle caratteristiche geografiche e dell’influenza delle attività turistiche.
I depuratori scelti:
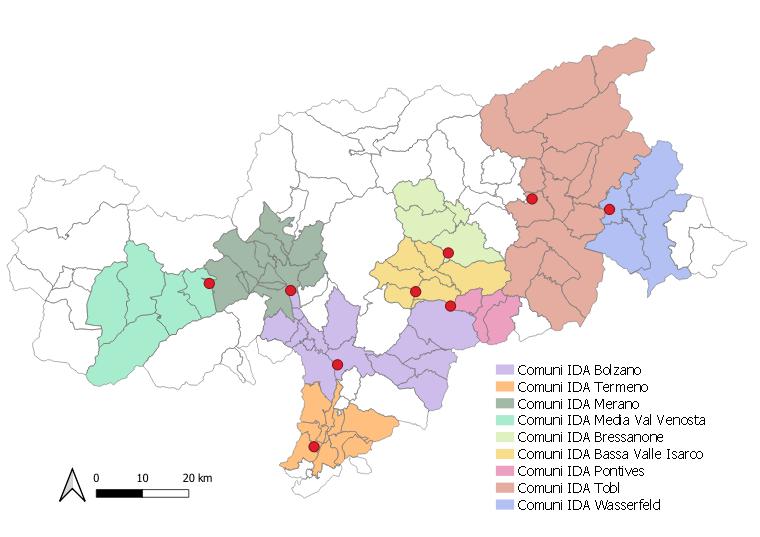
| Impianto depurazione acque reflue urbane (IDA) | Zone servite | Capacità in a.e. (abitanti equivalenti) | Abitanti residenti |
|---|---|---|---|
| IDA Bolzano | Bolzano (senza San Giacomo), Appiano (senza Monticolo), Terlano, Nalles, Gargazzone, Postal, Cornedo all’Isarco, Renon (Signato, Sill, Campodazzo, Unterplatten), San Genesio (senza Avigna e Valas), Tesimo, Castelrotto (Castelrotto, Siusi e Kompatsch), Andriano, Tires, Fié, Nova Levante e Nova Ponente (Val d’Ega) |
450.000 | 144.382 |
| IDA Termeno | Caldaro, Termeno, Ora, Egna, Montagna, Cortaccia, Trodena, Aldino, Monticolo, Vadena (Castel Varco, Campi al Lago e zona Monte) |
138.000 | 24.778 |
| IDA Pontives | Selva di Val Gardena, S: Cristina Val Gardena, Ortisei, Castelrotto (Oltretorrente, Saltria, Bulla), Lajon (Pontives) | 75.000 | 10.410 |
| IDA Media Val Venosta | Lasa, Silandro, Laces, Martello, Castelbello/Ciardes | 40.300 | 17.705 |
| IDA Bressanone | Bressanone, Varna, Naz-Sciaves, Fortezza, Campo di Trens (senza Mules), Velturno (Ziggler) | 65.000 | 31.718 |
| IDA Tobl | Rasun Anterselva, Valdaora, Perca, Predoi, Valle Aurina, Campo Tures, Selva Molini, Gais, Brunico, S. Lorenzo, La Valle S. Martino in Badia, Marebbe, Falzes, Badia (Pescol, Puntac) | 150.000 | 49.080 |
| IDA Wasserfeld | Braies, Dobbiaco, Monguelfo, Valle di Casies, Villabassa | 58.000 | 10.170 |
| IDA Bassa Val Isarco | Ponte Gardena, Barbiano, Villandro, Chiusa, Velturno, Lajon, Funes | 45.000 | 38.223 |
| IDA Merano | Naturno, Plaus, Parcines, Lagundo, Merano, Marlengo, Cermes, Lana (zona artigianale), Riffiano, Caines, Tirolo, Scena, Avelengo, Saltusio, S. Martino in Pass. (Saltusio, Quellenhof), S. Leonardo in Passiria (Sant'Orsola) |
364.000 | 69.600 |
.
Nelle acque reflue sono presenti quantità molto elevate di cellule di batteri e di virus, che popolano il tratto intestinale umano e che vengono espulsi con le feci. Per poter effettuare le analisi è necessario concentrare il campione in laboratorio per estrarre tutto il materiale genetico in esso presente. Il volume iniziale del campione prelevato è di 50 ml, che corrisponde ad una grande provetta, da cui si ottiene il materiale genetico concentrato in 0,1 ml (una goccia). In questa miscela di DNA ed RNA molto eterogenei di provenienza batterica, virale, umana si cercano alcune sequenze geniche dell’RNA(1) relative a proteine specifiche del Coronavirus SARS-CoV-2 attraverso la tecnica della real-time RT-PCR(2). L’intensità del segnale è in correlazione con la concentrazione dell’RNA, e quindi del virus in circolazione tra la popolazione.
Nota 1: L’RNA è il materiale genetico del SARS CoV-2, che contiene tutte le informazioni per la moltiplicazione del virus nelle cellule del suo ospite.
Nota 2: La real-time RT-PCR è una tecnica che consente di sintetizzare una molecola di DNA a doppio filamento a partire da uno stampo a RNA (trascrizione inversa, RT). Successivamente il DNA è amplificato da reazioni a catena di un enzima chiamato DNA-polimerasi. Dopo ogni ciclo di amplificazione, la quantità DNA raddoppia, diventando “visibile” allo strumento e quantificabile in tempo reale durante la reazione. Il corredo genetico del SARS- CoV-2 è un filamento di RNA, per questo motivo è necessario il passaggio di retrotrascrizione.
- Trasporto dei campioni dal punto di prelievo al laboratorio, in condizioni di temperatura refrigerata.
-
Aggiunta ai campioni di un virus di controllo per verificare l’efficienza di recupero del materiale genetico dal campione.
-
Concentrazione del campione da 50 ml a 0,1 ml.
-
Estrazione del materiale genetico (RNA).(1)
-
Amplificazione (moltiplicazione) in RT-qPCR(2) del gene ORF-1ab (nsp14) del virus SARS-CoV-2 e amplificazione parallela del virus di controllo per il calcolo dell’efficienza di estrazione.
-
Esito: presenza/assenza di SARS-CoV2 nei campioni e quantificazione relativa.
-
Elaborazione dei dati entro 48h dall’arrivo dei campioni in laboratorio.
Nota1: L’RNA è il materiale genetico del SARS CoV-2, che contiene tutte le informazioni per la moltiplicazione del virus nelle cellule del suo ospite.
Nota 2: La real-time RT-PCR è una tecnica che consente di sintetizzare una molecola di DNA a doppio filamento a partire da uno stampo a RNA (trascrizione inversa, RT). Successivamente il DNA è amplificato da reazioni a catena di un enzima chiamato DNA-polimerasi. Dopo ogni ciclo di amplificazione, la quantità DNA raddoppia, diventando “visibile” allo strumento e quantificabile in tempo reale durante la reazione. Il corredo genetico del SARS- CoV-2 è un filamento di RNA, per questo motivo è necessario il passaggio di retrotrascrizione.
- Nessun dato può essere interpretato da solo: tutti i dati relativi ad un singolo campionamento devono essere messi in relazione ai dati precedenti e a quelli successivi (dinamica della curva), perché per sua natura il campionamento potrebbe non essere rappresentativo.
- Non è possibile dalle analisi del refluo stabilire se il segnale debba essere attribuito ai nuovi infetti o a persone che hanno già passato la fase critica, ma che continuano ad emettere il virus.
- L’analisi dei reflui può essere utilizzata come early-warning per la sorveglianza del SARS-COV-2, come possibile strumento per il controllo della circolazione del virus nella popolazione
- L’analisi dei reflui può essere utile a monitorare la diffusione del virus, laddove il sistema di tracciamento dei positivi non riesca a stare al passo con il numero crescente dei contagi.
- L’analisi dei reflui può confermare se le misure di contenimento della circolazione del virus sono efficaci, con l’abbassamento dell’intensità del segnale.
- La curva della concentrazione dell’RNA(1) di SARS-CoV-2 nei reflui anticipa la curva dei cittadini, che afferiscono al singolo depuratore, positivi al virus di un periodo variabile tra i 4 e i 14 giorni.
Nota 1: L’RNA è il materiale genetico del SARS CoV-2, che contiene tutte le informazioni per la moltiplicazione del virus nelle cellule del suo ospite.
Epidemiologia basata sulle acque reflue
Grazie alle informazioni ottenute analizzando i reflui urbani è possibile prevedere l’andamento della curva epidemica: all’inizio dei focolai epidemici il materiale genetico del virus è già rilevabile nelle acque reflue prima della notifica dei casi clinici. Quando il numero dei contagi è elevato, la presenza del materiale genetico di CoV-2 nelle acque aumenta, successivamente cala in maniera graduale dopo che le persone sono guarite, perché l’emissione del virus con le feci può continuare fino a diverse settimane dopo la scomparsa dei sintomi. Il rilevamento dell’RNA(1) virale nei reflui restituisce un buon quadro del trend in salita o in discesa della curva, fornendo informazioni preziose in tempo reale sulla circolazione del virus. La sorveglianza ambientale sui virus in acque reflue – o l’epidemiologia basata sulle acque reflue - è pertanto uno strumento di prevenzione e controllo di focolai epidemici, con i seguenti vantaggi:
-
Monitoraggio indiretto, anonimo e non invasivo della popolazione di una determinata zona attraverso la rilevazione del materiale genetico del virus nelle acque reflue.
-
informazioni sulla diffusione e circolazione del virus, con indicazioni sul trend nel corso del tempo.
-
Informazioni a supporto di decisioni su eventuali misure di restrizione, anche limitate alle zone più interessate alla circolazione del virus. I dati dell'epidemiologia delle acque reflue. possono essere utilizzati in combinazione con i dati epidemiologici della Sanità.
-
capacità predittiva in caso di eventuali ulteriori ondate di contagi.
Nota 1: L’RNA è il materiale genetico del SARS CoV-2, che contiene tutte le informazioni per la moltiplicazione del virus nelle cellule del suo ospite.
Acque reflue: le nuove opportunità della sorveglianza ambientale
(Produzione: agosto 2023)
Il virus nelle acque di scarico è attivo e infettivo?
La presenza del materiale genetico indica che il virus è presente nelle acque reflue, ma non ci sono evidenze che il SARS-CoV-2 possa sopravvivere nelle acque reflue o in altre matrici acquatiche. Per essere infettivo, il virus deve preservare intatti l’involucro che racchiude l’RNA(1) (il capside virale), la membrana esterna che lo avvolge e le proteine per il riconoscimento della cellula ospite da infettare. La presenza nelle acque di scarico di sostanze detergenti, di materia organica e delle cellule batteriche contribuisce a rovinare e degradare la membrana esterna del virus, inattivandolo e riducendo la sua capacità di infettare le cellule umane. Il SARS-CoV-2 si disattiva molto più rapidamente rispetto ai virus enterici a tipica trasmissione acquatica, come il norovirus, il rotavirus e il virus dell'epatite A. Attualmente non ci sono prove di trasmissione del SARS-CoV-2 nelle acque reflue o di infezione, poiché nelle acque reflue sono presenti solo frammenti dell'RNA virale, rendendo l'analisi delle acque reflue adatta al monitoraggio epidemiologico ambientale.
Nota 1: L’RNA è il materiale genetico del SARS CoV-2, che contiene tutte le informazioni per la moltiplicazione del virus nelle cellule del suo ospite.
Contatto: Laboratorio biologico


